肺癌是最常见的癌症,目前,CT可用于帮助医生在早期阶段检测肺癌。 在许多情况下,识别肺癌的诊断取决于医生的经验,这可能会忽略一些患者并导致一些问题。 在许多医学影像诊断领域,深度学习已被证明是一种流行且有效的方法。 本文主要基于LIDC-IDRI这一公开数据集,对其进行了肺结节的提取,并利用CNN对其分类训练,从而辅助医生作出判断。
由于篇幅较长,将分为2篇博客,这篇主要介绍数据处理,即肺结节的提取。
数据集
数据集采用为 LIDC-IDRI (The Lung Image Database Consortium),该数据集由胸部医学图像文件(.dcm)(如CT、X光片)和对应的诊断结果病变标注(.xml)组成。数据是由美国国家癌症研究所(National Cancer Institute)发起收集的,目的是为了研究高危人群早期癌症检测。
该数据集中,共收录了1018个研究实例。对于每个实例中的图像,都由4位经验丰富的胸部放射科医师进行两阶段的诊断标注。在第一阶段,每位医师分别独立诊断并标注病患位置,其中会标注三中类别:
- $>=$3mm的结节
- $<$3mm的结节
- $>=$3mm的非结节
在随后的第二阶段中,各位医师都分别独立的复审其他三位医师的标注,并给出自己最终的诊断结果。这样的两阶段标注可以在避免forced consensus的前提下,尽可能完整的标注所有结果。
图像信息(.dcm)
图像文件为Dicom格式,是医疗图像的标准格式,其中除了图像像素外,还有一些辅助的元数据如图像类型、图像时间等信息。
一张CT图像有 512x512 个像素点,在dicom文件中每个像素由2字节表示,所以每张图片约512KB大小。
目前测试一共1012个病例数据,对于每个实例,可以看为一个三维矩阵D(slicer rows cols), slicer表示切片的个数(对应每个病例的.dcm文件数),rows和cols分别表示图片的行数和列数(默认为512)。
eg: 对于病例LIDC-IDRI-0001,即为$133 \times 512 \times 512$的矩阵,一共133张切片,每张大小$512 \times 512$。
查看dcm文件:
- 通过pip或者Anaconda安装pydicom模块,该模块是python专门用来处理dicom格式文件的库。
- 通过软件MicroDicom viewer
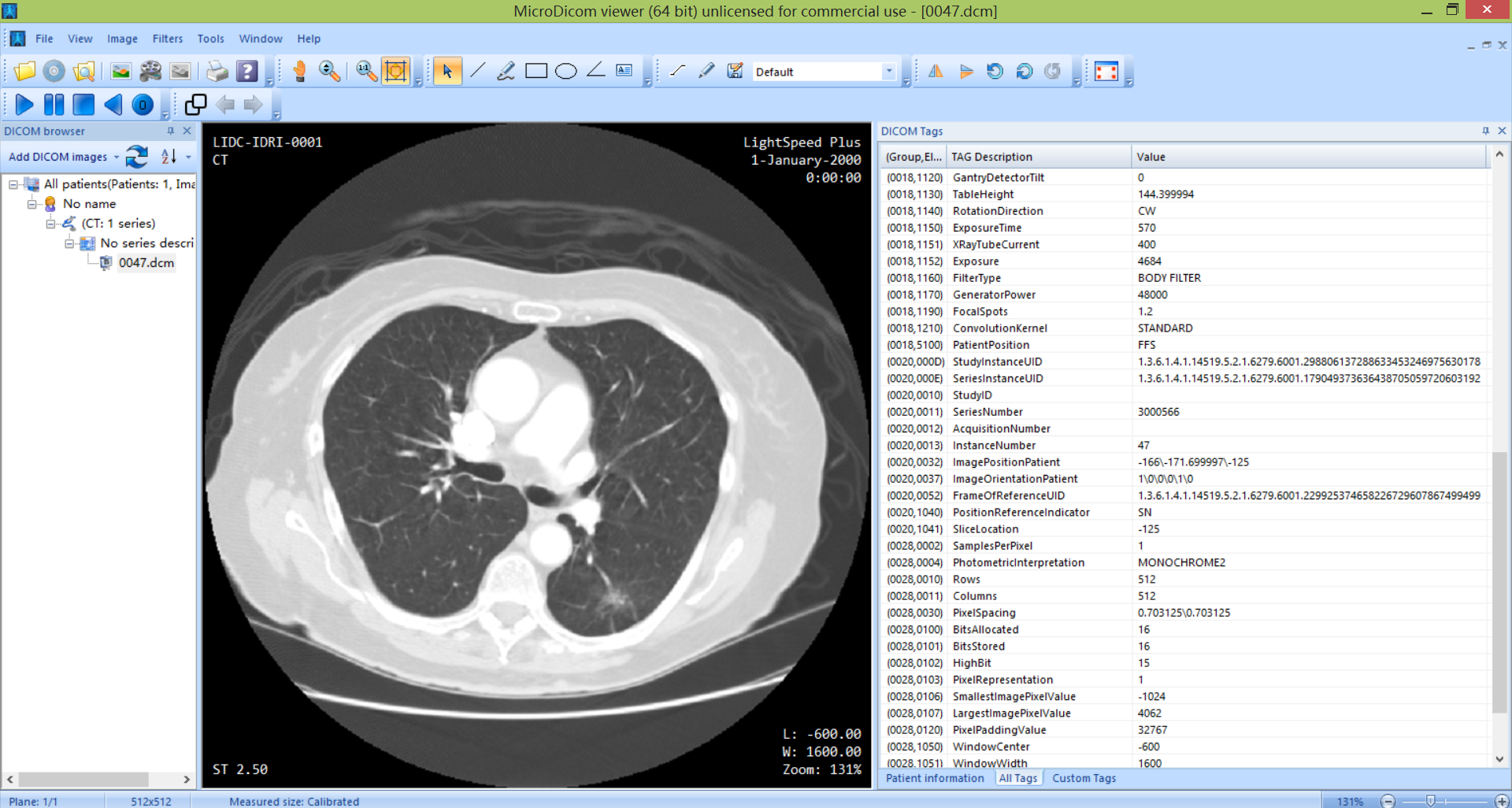
通过上面2种方式,我们可以看出dicom文件中包含了一些图像信息(SOP Instance UID、Study Instance UID,Series Instance UID······)
SOP Instance UID:用于唯一区分每一张dcm切片
Study Instance UID: 每个病例对应的检查实例号
Series Instance UID: 不同检查对应的序列实例号
注释信息(.xml)
Xml文件中包含放射科医生对病人CT图像中疑似肺结节的标注信息,主要分为三类:
- 结节(3mm-33mm):包含结节的特征信息(characteristics)、结节的完整轮廓(roi)
- 结节(<3mm):只显示结节的近似三维重心,若不透明则不标记
- 非结节(>3mm):只显示其近似的三维重心,指出非结节连接区域
Xml文件大体结构图如下:
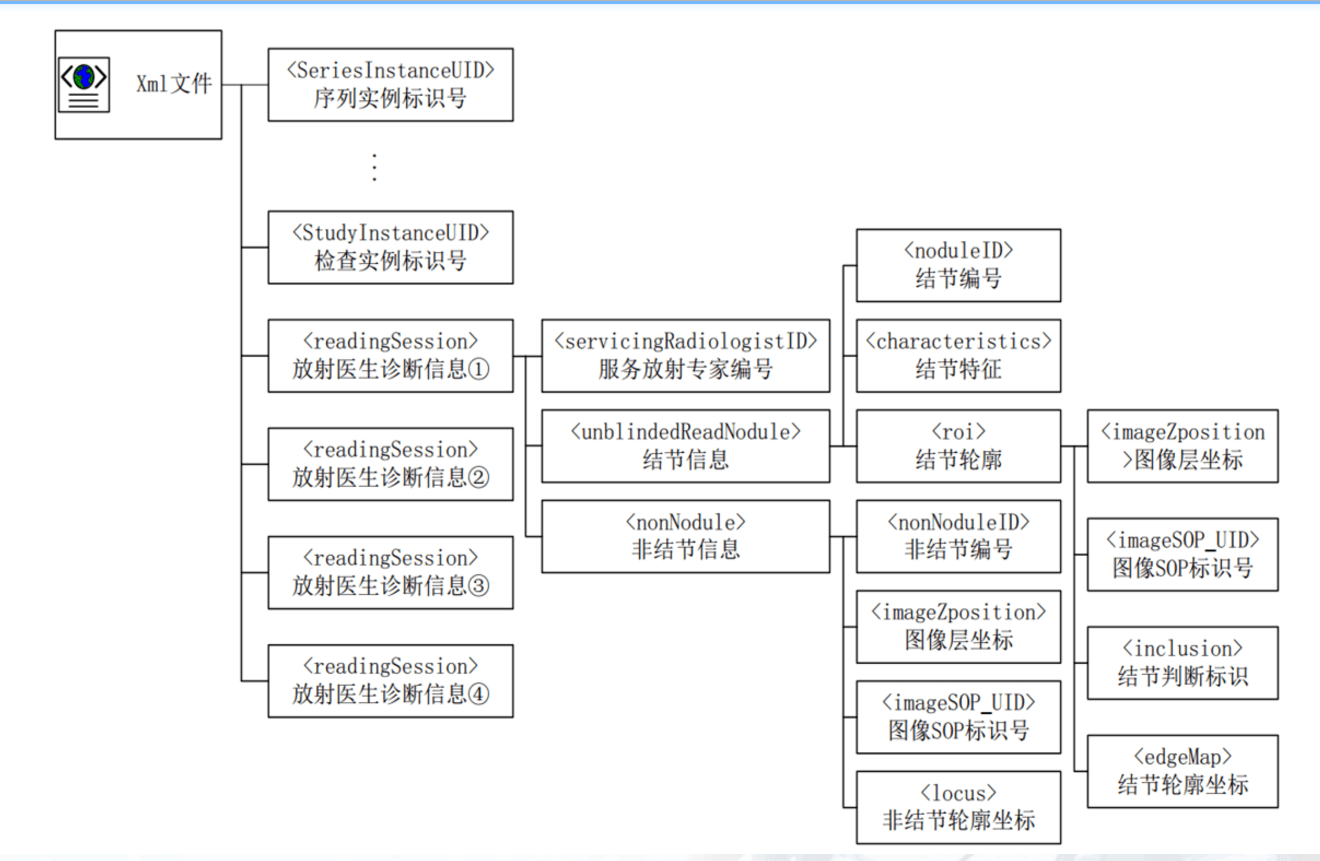
其中对于3mm—33mm结节的characteristics,包含了如下信息:
1) Subtlety:检测难度(1-5级,1最难,5最明显)
2) internalStructure:内部结构(4种,软组织、液体、脂肪、空气)
3) calcification:钙化(6种情况)
4) sphericity:球形度(5种程度,但只明确3种)
5) margin:边缘(5种程度)
6) lobulation:分叶征(5种情况,但只明确2种)
7) spiculation:毛刺征(5种情况,但只明确2种)
8) texture:纹理(5种情况,但只明确3种)
数据预处理
本部分主要做的工作是分割肺实质,提取肺结节。
图像存储格式转换
原始数据集的图像信息是以dcm格式存储的,但通常我们用作训练数据输入网络的图像大多是jpg或者png格式,所以为了方便以后的训练,我们首先要将原始图像转为jpg格式或者png格式存储,在这里我们是转为jpg格式存储的。
此处测试共包含1012个病例,每个病例包含约100—300个dcm文件,我们使用MicroDicom viewer软件对其进行批量转换。
以LIDC-IDRI-0001 为例:
原始数据
转换后
将原始数据转换为jpg格式的图片后,下面我们会利用matlab编写函数分割肺实质,提取肺结节,主要包含下面6个函数:
find_files(): 递归的遍历文件目录fengefeishizhi(): 分割肺实质readxml(): 读取标注信息(.xml)文件readdicom()存储标注信息中的肺结节信息,方便后面提取jianqieimage(): 剪切肺结节jianqie(): 根据肺结节的轮廓信息将其剪切出来,存为图片
分割肺实质
将图像转换为jpg格式存储后,我们还要对数据进一步处理。由于我们最终是以肺结节图像作为训练数据输入网络,那么CT图像中除肺部以外的信息是无用的,所以我们要将肺实质分割出来。主要用到1个函数: fengefeishizhi()。
代码如下:
1 | clear all; |
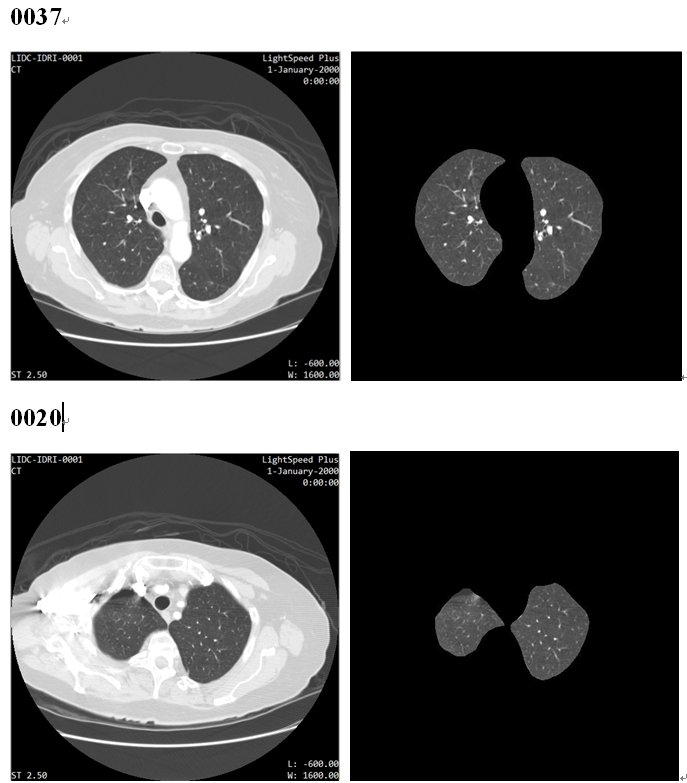
以LIDC-IDRI-0001 中的部分切片为例,其中左侧为原始CT图像,右侧为分割肺实质后的图像,效果图如下:
读取标注信息并存储
我们从医生的标注信息文件(.xml)读取肺结节的位置信息和良恶性程度,然后存储到对应的xls表中。主要用到2个函数: readxml()和readdicom()
代码如下:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72function [num_mal,sop_text,max_min_xy]=zl_readxml(xml_path)
% % function [sop_text,max_min_xy]=zl_readxml(xml_path)
% clear all
% clc
%xml_path = 'H:\肺结节\数据\LIDC-IDRI\900-300\LIDC-IDRI\LIDC-IDRI-0060\1.3.6.1.4.1.14519.5.2.1.6279.6001.203745372924354240670222118382\1.3.6.1.4.1.14519.5.2.1.6279.6001.463214953282361219537913355115\191.xml';
%% 跳转到内层标签unblindedReadNodule
docNode = xmlread(xml_path); %读取XML文件返回一个文件模型节点*
document = docNode.getDocumentElement();
readingSession = document.getElementsByTagName('readingSession'); %返回与给定的元素所有子节点的Nodelist对象*
%% 最后返回的三个值
%% 最后返回的三个值
num_mal = []; %每个结节的恶性度和属于该类别的图片的数量
sop_text = { }; %每个图片的标号
max_min_xy = []; %每个图像中肺结节的x和y的最小值和最大值
sop_num = 0; %总结节个数?*
%%
for r = 0:readingSession.getLength()-1
unblinded_nodule = readingSession.item(r).getElementsByTagName('unblindedReadNodule'); %unblindedReadNodule一个节点标记,<unblindedReadNodule>节点数据包括在</unblindedReadNodule>*
for u = 0 : unblinded_nodule.getLength()-1
roi = unblinded_nodule.item(u).getElementsByTagName('roi'); %item() 方法可返回节点列表中处于指定索引号的节点。*<roi>结节轮廓</roi>*
mal = unblinded_nodule.item(u).getElementsByTagName('malignancy'); %<malignancy>结节恶性度</malignancy>*
%如果xml文件中没有malignancy或者roi标签直接跳过
if isempty(roi.item(0))
continue;
end
if isempty(mal.item(0))
continue;
end
Num_roi = roi.getLength(); %该类别的图片的数量
mal_int = str2num(char(mal.item(0).getTextContent()));
num_mal = [num_mal();mal_int,Num_roi];
for i = 0 : Num_roi-1 %遍历*
sop_id = roi.item(i).getElementsByTagName('imageSOP_UID'); %图片编号*
sop_text{sop_num + i + 1} = char(sop_id.item(0).getTextContent()); %数组*
edgeMap = roi.item(i).getElementsByTagName('edgeMap'); %边界*
xy = [];
for j = 0 :edgeMap.getLength()-1 %获得坐标*
xCoord = edgeMap.item(j).getElementsByTagName('xCoord');
xCoord_int = str2num(char(xCoord.item(0).getTextContent()));
yCoord = edgeMap.item(j).getElementsByTagName('yCoord');
yCoord_int = str2num(char(yCoord.item(0).getTextContent()));
xy=[xy();xCoord_int,yCoord_int];
end
%找到结节轮廓*
if edgeMap.getLength()==1
max_min_xy = [max_min_xy();xy,xy];
continue;
end
[maxr,max_index] = max(xy);
[minr,min_index] = min(xy);
max_min_xy = [max_min_xy();minr,maxr];
end
sop_num = sop_num + Num_roi; %总个数
end
if isempty(num_mal)
continue;
end
num_mal = [num_mal();0,0]; %扩展维数*
end
end
上述是辅助函数,提取函数如下所示:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84clear;
clc;
%% Each treatment 100 or 200 %处理数据导入到表格中
%LIDC_path = 'E:\zhaolei\深度学习\肺结节\400-499\LIDC-IDRI\'; 原路径
LIDC_path = 'D:\MATLAB\tiqu\LIDC-IDRI';
%IDRI_path = 'H:\肺结节\数据\LIDC-IDRI\'; 原路径
%XLS_path = 'H:\肺结节\数据\excel\excel_all'; 原路径
IDRI_path = 'D:\MATLAB\tiqu\LIDC-IDRI';
XLS_path = 'D:\MATLAB\tiqu\xls';
IDRI_child_path = dir(IDRI_path); %打开文件目录并返回文件结构体*
num_IDRI_child = size(IDRI_child_path); %返回列和行数的数组*
%for n = 8 :num_IDRI_child 原版 %为啥从8开始???
for n = 3 :num_IDRI_child %非原版
% child_idri_path = [IDRI_path,IDRI_child_path(n).name];原版(可能有错)
child_idri_path = [IDRI_path,'\',IDRI_child_path(n).name]; %非原版
child_idri_path_temp = dir(child_idri_path); %打开文件*
LIDC_path = [child_idri_path,'\',child_idri_path_temp(3).name]; %文件目录*
LIDC_child_path = dir(LIDC_path); %打开
num_child = size(LIDC_child_path); %返回文件的列和行数的数组*
for i = 3 : num_child(1) %从3开始(前两个是. ..)
%% find dicom file list
child_path = [LIDC_path,'\',LIDC_child_path(i).name]; %一步步打开文件夹
child_path_temp = dir(child_path);
child_path1 = [child_path,'\',child_path_temp(3).name];
child_path_temp = dir(child_path1);
%xml_path = [child_path1,'\',child_path_temp(3).name];
xml_path = [child_path,'\'];
%获取单个文件夹中的dicom和xml文件
dcm_files = find_files(xml_path, '.dcm'); % 获得文件列表
xml_files = find_files(xml_path, '.xml');
xml_path = char(xml_files);
[num_mal,sop_text,max_min_xy]=zl_readxml(xml_path); %函数调用
% num_mal = []; %每个结节的恶性度和属于该类别的图片的数量
% sop_text = { }; %每个图片的标号
% max_min_xy = []; %每个图像中肺结节的x和y的最小值和最大值
sop_num = size(sop_text); % 获得行列数,行:? 列:图片数*
mal_num = size(num_mal); %行: 图片数?*
dcm_number = [ ]; %图片编号*
%??
if sop_num(2)>mal_num(1) %要根据他们两个的差值来决定补多少个0
for m = 1 : sop_num(2)-mal_num(1)
num_mal = [num_mal();0,0]; %添加扩展维度*
end
end
if sop_num(2)< mal_num(1)
for m = 1 : mal_num(1) - sop_num(2) % 只有数据维度一样才能被写入到文件中!所以少的要补上四个0
dcm_number= [dcm_number;0]; %添加扩展维度
max_min_xy = [max_min_xy;0,0,0,0]; %添加扩展维度
end
end
%??
%% Get the number and file name of the image In a single folder
for md = 1 : sop_num(2) %???
dcm_number= [dcm_number;0];
end
for j = 1:numel(dcm_files) %遍历文件
dicomInformation = dicominfo(dcm_files{j}); %存储图片信息
instance = dicomInformation.SOPInstanceUID;
imagenum = dicomInformation.InstanceNumber;
% Make sure that the StudyInstanceUID matches that found in
% the XML annotations
for s = 1 : sop_num(2) %对比
if strcmpi(instance,sop_text(1,s))
dcm_number(s) = imagenum; %编号???*
end
end
end
total = [num_mal,dcm_number,max_min_xy];
if isempty(total)
continue;
end
child_path = [XLS_path,'\',LIDC_child_path(i).name]
xlswrite(child_path,total); %导入到表格中 2017/4/10
end
end
提取肺结节
读取到肺结节的位置信息和良恶性程度后,我们要根据该信息提取肺结节。主要用到2个函数: jianqieimage()和jianqie()
代码如下:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46function zl_jianqie(img_path,dir,times,size_center,col4xy)
%dir 为患病可能程度,col4xy为剪切区域
train_path = 'I:\肺结节\数据\result2\train23jpg\';
result_name = [img_path(24:37),'_',char(num2str(dir)),'_',char(num2str(times)),img_path(38:46)];
train_path = [train_path,char(num2str(dir)),'\',result_name]; %剪切路径*
img=imread(img_path); %读取图片文件*
img1=imcrop(img,col4xy); %返回图像的一个裁剪区域* I2=imcrop(I,[a b c d]);%利用裁剪函数裁剪图像,其中,
%(a,b)表示裁剪后左上角像素在原图像中的位置;c表示裁剪后图像的宽,d表示裁剪后图像的高
%% 分割肺结节实质
img1_size = size(img1);
min(min(img1)); % 找到最小值,最大值
max(max(img1));
t=graythresh(img1); %使用最大类间方差法找到图片的一个合适的阈值threshold
C=im2bw(img1,t); %转换为二值图像*
D=imfill(C,4,'holes');%对二值化后的图像填充肺实质
if dir >=4 %大概率为肺癌*
FMask=bwareaopen(D,10); % 除二值图像中面积小于10的对象
D = FMask;
end
total = 0;
for i = 1:img1_size(1) %行数
for j = 1:img1_size(2) %列数
if D(i,j) == 0 %二值图像当值为0时 (黑色)
img1(i,j) = 0;
end
if D(i,j) == 1 %二值图像当值为1时 (白色)
if ~(i > size_center(1) && j > size_center(1)&& j < size_center(1) + size_center(3)&& i < size_center(1) + size_center(3)) %不在范围内*?
img1(i,j) = 0; %取为黑色*
end
end
end
end
%% 保存图片
for m = 1:img1_size(1)
for n = 1:img1_size(2)
if img1(m,n) == 0 %黑色元素点个数*
total = total + 1;
end
end
end
if total ~= img1_size(1)*img1_size(2) %如果不全是黑*
imwrite(img1,train_path); %存入图片*
end
end
1 | clear; |
通过上面的函数,我们可以将肺结节提取出来,并按照良恶性程度分类存储。
部分示例如下:

其中1-5表示肺结节的良恶性程度,5表示恶性可能性最大,1表示恶性可能性最小。
注意:3表示不确定是否为肺结节,良恶性程度也不确定。
至此,我们的数据预处理结束了,分类训练见下一篇博文。